微生物测序
微生物组(Microbiome)是指一个特定环境或生态系统中全部微生物及其遗传信息的集合, 蕴藏着极为丰富的微生物资源。全面系统地解析微生物组的结构和功能, 将为解决人类面临的能源、生态环境、工农业生产和人体健康等重大问题带来新思路
细菌完成图(三代测序)
基因组是阐释生命现象和揭示生命规律的重要手段,三代PacBio HiFi测序模式可产生既兼顾长读长,又具有高精度的测序结果,贝瑞基因HiFi细菌完成图测序,即利用PacBio HiFi测序模式对细菌物种进行基因组de novo组装,从而获得该细菌种完整基因组序列,该技术大大提升了细菌基因组组装的完整性和准确性,可真正实现细菌0 gap组装。
真菌精细图(三代测序)
三代真菌精细图,即采用PacBio测序平台,利用高深度三代数据进行真菌基因组de novo组装,获得真菌基因组序列信息的一种技术手段。真菌基因组一般在10M-150M,有多核和杂合等现象,利用三代测序技术的优势破译真菌基因组,能克服二代测序对高GC、高重复、高杂合位置检出不准确的现象,得到更加完整和均匀的基因组,结合HiC辅助组装技术可获得染色体水平全基因组序列图谱,此外,测序同时还可获得甲基化修饰位点信息。
全长16s(三代测序)
常规二代16S仅针对单个可变区或者连续两到三个可变区进行测序,而基于PacBio HiFi测序的全长16S(V1-V9)可获取微生物16S的全长序列,突破了二代测序读长较短的局限性,能够更好的实现微生物在种水平上的分类, 可获得物种分类、丰度、种群结构等更加深入和全面的信息。基于PacBio HiFi测序技术的全长16s测序为微生物多样性研究提供了强有力的技术手段。
宏基因组(二代测序)
二代宏基因组测序技术是依托二代测序平台以特定环境中的整个微生物群落作为研究的对象,不需要对微生物进行分离培养,提取环境微生物总DNA进行研究,进行微生物群体的物种分类、复杂度分析、群落结构、功能注释、样品间的物种或基因差异及物种间的代谢网络研究。与传统微生物研究方法相比,宏基因组测序技术规避了绝大部分微生物不能培养、痕量菌无法检测的缺点。
应用方向
- 精准医疗:探究肠道、皮肤、阴道等菌群环境对疾病的影响,寻找与代谢类疾病、消化类疾病、自身免疫性疾病等相关的biomarker及与癌症相关的治疗靶点;
- 药物代谢:探究功能食品、中药的体内代谢及药效机制;
- 传统发酵生产:关键风味物质溯源及控制发酵微生物菌种优化改良,及特殊功能菌株、基因挖掘、工程菌开发等;
- 畜牧渔养殖:研究肠道、瘤胃等微生物与动物繁殖、生长发育、营养健康、免疫和疾病治疗等的互作关系、对环境的响应及对生产的影响;
- 大农业:研究根系微生物对植物生长、抗逆的影响,指导种植方式;
- 环境领域:研究特定环境下微生物研究,解决有机肥发酵处理、污水治理、石油降解、水体及海洋环境等环境相关问题;
案例解析
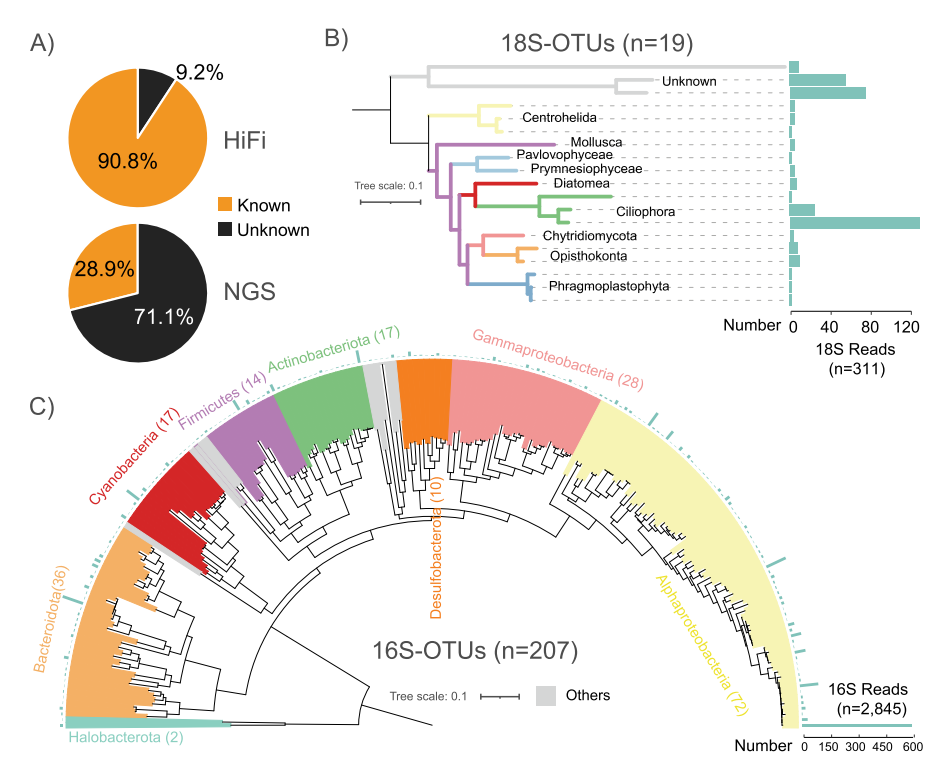
基于HiFi reads的分类分辨率。(A) HiFi和NGS读数具有(已知)/不具有(未知)分类信息的百分比。(B和C)基于19个全长18S-OTUs (B)和207个全长16S-OTUs (C)的系统进化树
利用HiFi宏基因组测序技术改进西藏盐湖沉积物中宏基因组组装和病毒组装
该文通过应用长读长测序技术和短读长测序技术对青藏高原盐湖沉积物样品进行测序,评估了不同组装策略在宏基因组组装和病毒组装中的性能。28.9% 的 NGS 读取可以分配到已知的分类等级,而HiFi数据的读取分配率为90.8%,大约是NGS的三倍。HiFi reads的物种级别分配率为67.19%,而短读长的相应比率仅为5.59%,说明HiFi reads在分类方面的强大能力。总共鉴定出17个最小相对丰度大于0.1%的门,大大超过了NGS技术的检索性能。这为复杂环境样品的物种组成研究,特别是对真核微生物的了解提供了便利。
参考文献:Tao Y, Xun F, Zhao C, Mao Z, et al. Improved Assembly of Metagenome-Assembled Genomes and Viruses in Tibetan Saline Lake Sediment by HiFi Metagenomic Sequencing. Microbiol Spectr. 2023 Feb 14;11(1):e0332822.

